整合RNA和蛋白质组学数据识别多形性胶质母细胞瘤表面组特征
Abstract
本文目的是发掘GBM相关的表面组基因以及识别关键的具有发展潜力的细胞表面基因作为GBM分子标志物用于治疗。主要利用TCGA GBM RNA-Seq数据以及GTEx正常脑皮层数据识别在GBM中显著失调的表面组基因,本文还综合分析了转录组学,蛋白组学数据来区分具有高置信度的GBM表面组特征。
Background
多形性胶质母细胞瘤(GBM)是最常见的且致死率高的成人中枢神经系统肿瘤。尽管近些年已经开发了很多治疗方法,但是该疾病的中位生存期依旧较差。GBM患者的平均生存时间期望只有个月且五年内的生存期低于3%。目前标准的GBM治疗方案通常是在最大程度上安全的手术切除的基础上,伴随着放化疗的治疗。然而该病具有高复发率和其肿瘤内很高的异质性导致治疗的预后很差。因此,需要开发更好且有效的治疗方案。
替莫挫胺是最常用的口服脱氧核糖核酸烷化(DNA-alkylating)药物,常用于在临床上GBM的化疗。常见的联合替莫挫胺和放射线疗法的标准化的Stupp治疗方案常用于治疗GBM。其他的GBM治疗方案有例如VEGF靶向的单克隆抗体贝伐单抗治疗、脱氧核糖核酸烷化药物或者检查点抑制剂治疗等。
细胞表面蛋白质或表面基因组是一个整合和转运细胞内外关键信号级联的信息阀门。同时,表面基因组也在肿瘤发生的支持和侵袭的关键进展中起重要作用。事实上,异常的表面基因组表达和活性常在许多癌症类型中观察到。因此,表面基因组在癌症的治疗靶向或生物分子标志物中具有很大的潜力。最近的一项研究表明,56%的细胞表面蛋白在GBM中具有不同的表达并且在血浆以及脑脊液中也可以观察到这一现象,表明表面组蛋白具有成为生物标志物的潜力。质谱仪分析显示,表面基因组在患癌脑细胞系中平均大小大于其他(部位)类型癌症。因此,GBM表面组基因可能是我们理解GBM发病机制和药物反应的关键,靶向这些基因可能解开GBM治疗中潜在“可药性”阶段。
目前,一个全面综合的GBM表面基因组图谱概述还没有完全定义。因此,本文旨在使用TCGA(GBM)和GTEx(normal brain)数据库的数据分析GBM表面组基因表达图谱的特征。由于TCGA正常脑组织样本较少,所以本文在两个数据库整合和完成了差异基因表达分析。为了进一步获得具有高置信度的GBM表面基因标志,我们使用PPI hub基因分析整合了GBM转录组和细胞表面蛋白质组的特征。总而言之,我们识别出了在GBM上调的表面组基因包括CD44,PTPRJ和HLA-DRA,这些基因在未来研究GBM发病机制的生物相关性可能有重要作用。
Result
TCGA和GTEx患者的特征
如表所示,对TCGA数据分别进行性别,治疗年龄以及治疗效果进行分组。
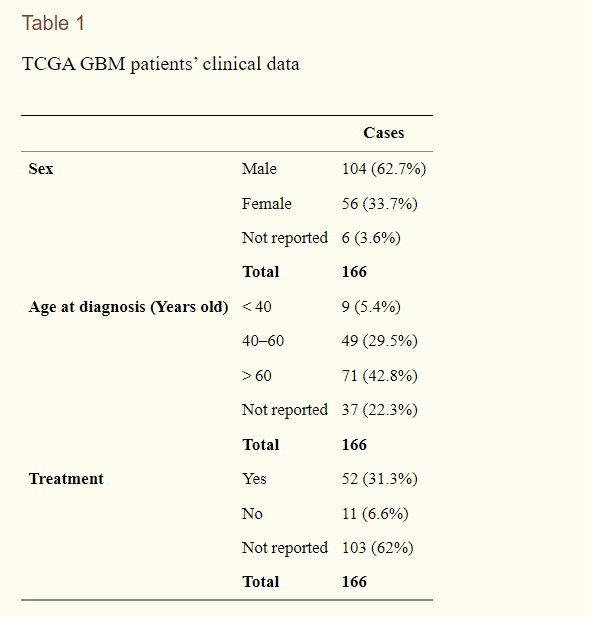
识别GBM的差异表达基因
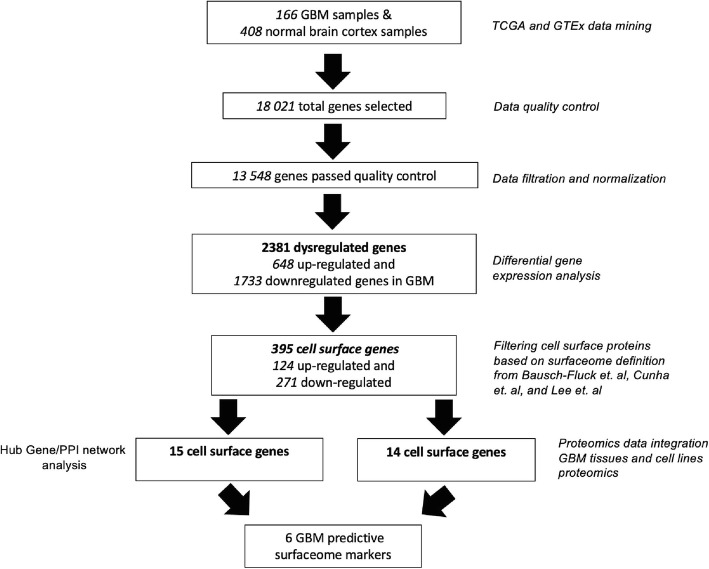
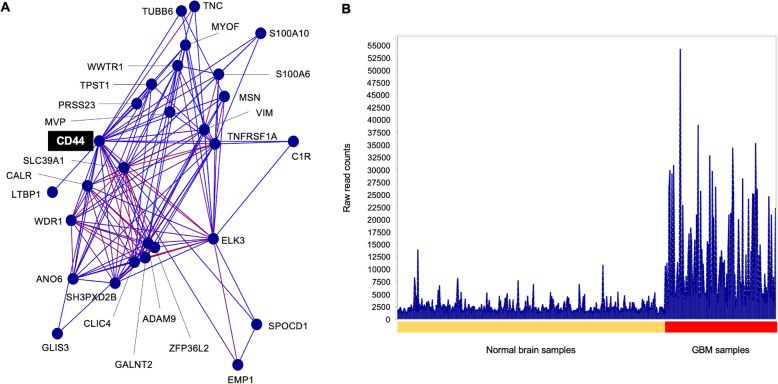
图1显示差异基因分析流程。由于大多数GBM病例都发现于大脑的幕上区域,文章仅选取了GTE中RNA-Seq表达谱中的皮质,正面皮质以及前刺铰接皮层。图2.A显示利用t-SNE分析反映了GBM肿瘤和正常脑组织read count 值的方向性。图2.A清晰地显示了表达谱中正常脑组织和肿瘤中表达模式的不同。最终,RNA表达数据中18021个基因有13548个基因通过了质控。通过设置差异基因的阈值(具体阈值见Methods),在GBM中找到了2381个DEGs,其中有648个基因上调,1733个基因下调(见图2.B)。
对DEGs的功能富集和分类
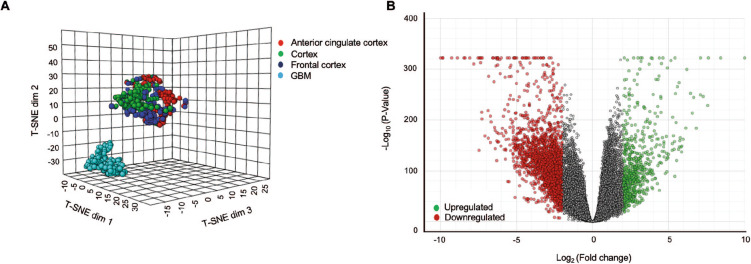
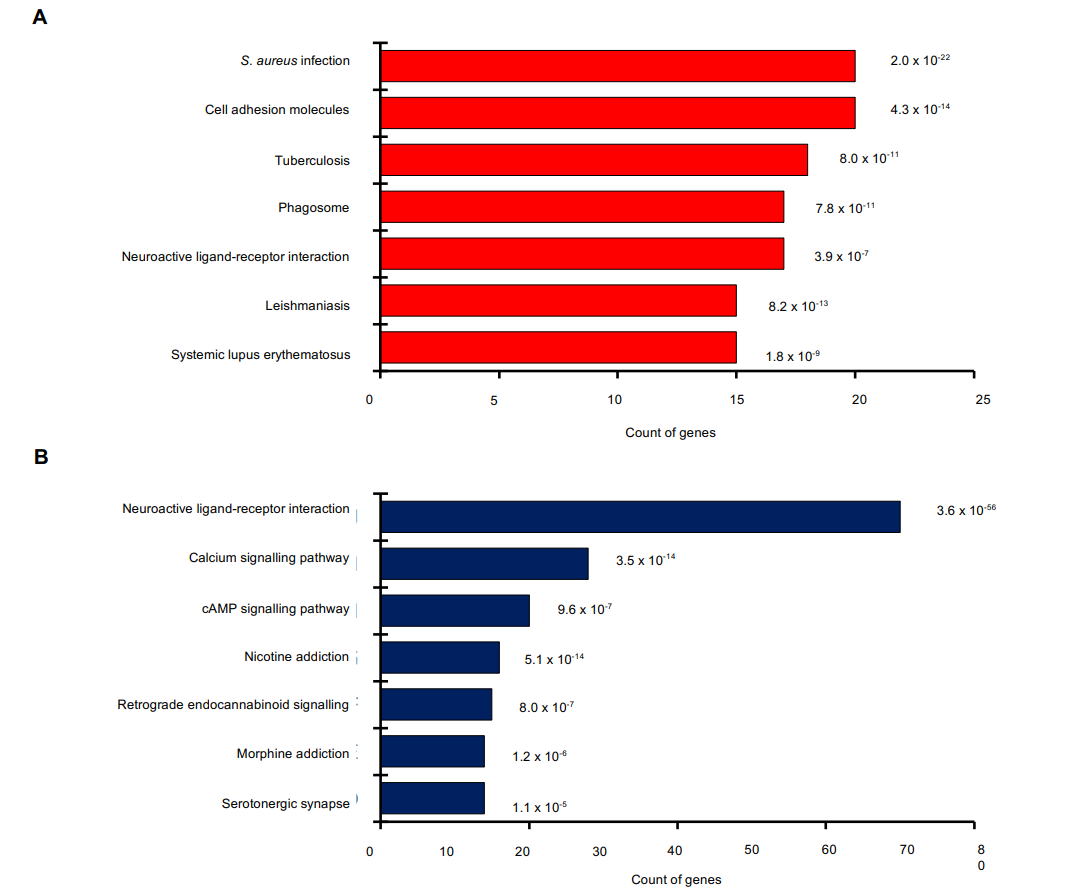
对上述结果找到的差异基因进行GO和KEGG的功能富集。对于上调/下调的差异基因,GO富集显示差异基因与细胞表面和膜相关蛋白相关联;KEGG富集显示,上调基因主要涉及癌症传播和细胞黏附相关的通路,下调基因主要涉及神经活性配体-受体交互和主要细胞信号传导相关的通路。(图S1.ALL)
识别GBM细胞表面抗原的候选分子
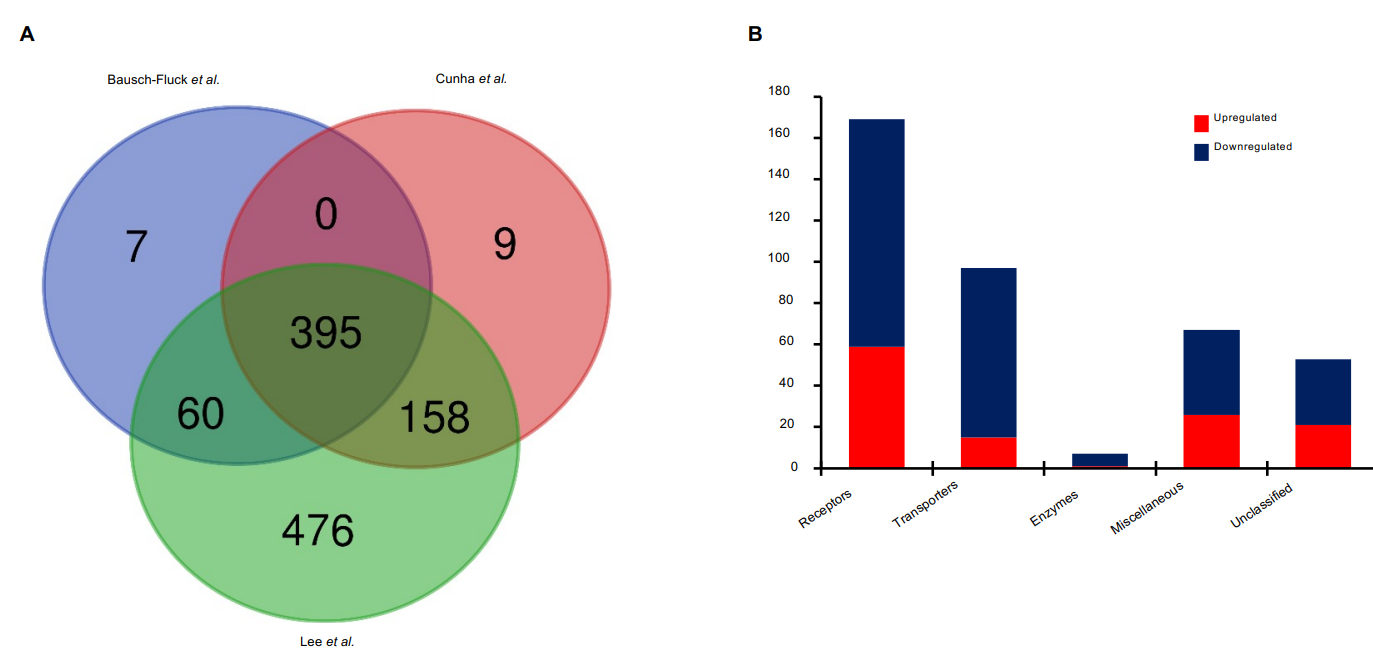
Bausch-Fluck, Cunha. and Lee.等人的研究通过不同的研究方法,已经获得了表面组差异基因的基因列表。对他们基因列表取交集,得到了395个共同表面组差异基因,其中有124个上调基因,271个下调基因(图S2.A)。根据Almén等人的研究,表面组可以分为以下五个亚型:受体型,运输型,酶型,混杂型和未定义型。将图A中的结果根据亚型分类,可以发现42.8%的GBM差异表达基因属于受体亚型(图S2.B)。KEGG分析GBM-富集表面蛋白识别到与免疫防御和传播相关的通路;而GBM-缺乏细胞表面基因富集到与神经活性韧带交互和主要信号传导相关通路(图S3.ALL)。这一结果与之前所做的差异基因富集相类似,表明表面基因组在控制GBM细胞活动中起到重要作用。
整合转录组和蛋白组数据识别GBM细胞表面标志
目前为止,我们已经
(i)使用转录组数据在GBM分类了所有DEGs。
(ii)在GBM中找到了细胞表面差异表达基因。
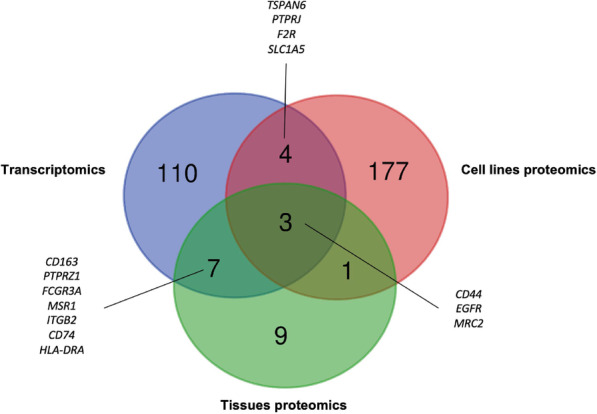
即使转录组分析基因提供了十分有效的信息用于发现的生物标志物,为了进一步深入探究生物标志物的可靠性,我们决定添加一层新的分析用于筛选具有更高置信度的GBM细胞表面标志。所以我们整合了上述的转录组数据和公开数据集中可访问的蛋白组数据。该整合分析可以用于细胞表面基因的预测和消除由于转录/翻译后产生的在mRNA和蛋白表达水平的差异(不一致)。因此,我们下载了大量公开数据集提供的大量的GBM组织和细胞系的质谱分析数据。我们假定GBM组织和细胞系拥有不同细胞表面库,并且我们因此可以对两者进行分层。
对五个细胞系样本的质谱分析显示了EGFR,CD44,PTPRJ,SLC1A5,F2R和TSPAN6蛋白的上调,而该结果与图F.3转录组结果一致。对于组织样本,我们只找到一个可访问的研究(Polisetty等),使用其数据找到了10个交叠基因:MRC2, FCGR3A, HLA-DRA, CD44, CD74, MSR1, CD163, EGFR, ITGB2, PTPRZ1(图F.3)。其中只有PTPRZ1基因表达上调,其余基因都为下调。以上,共有14个转录组,组织和细胞交叠基因。值得注意的是,质谱分析蛋白可能会由于蛋白溶解/隔离或其他内在变化影响质谱分析仪器对蛋白的敏感性和探测的能力。因此这些限制可能会导致预测结果的偏差。
表面基因组蛋白-蛋白交联网络分析和高置信度GBM细胞表面组标志物的优先级
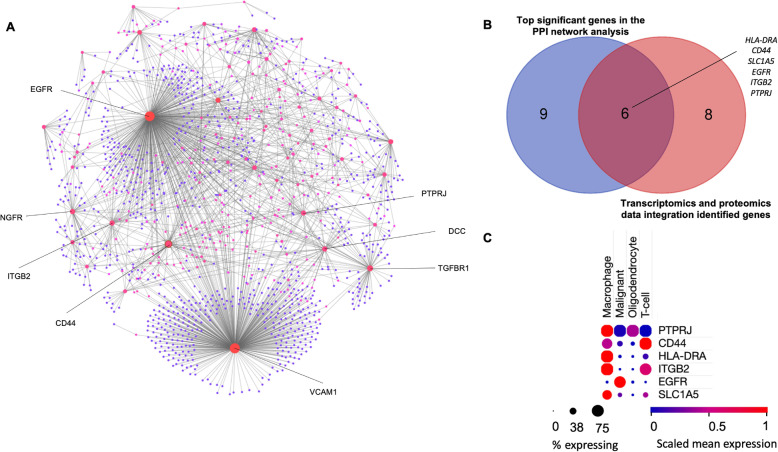
接下来,我们进一步使用蛋白-蛋白交联(PPI)网络分析GBM-富集细胞表面标志物。通过该分析可以帮助我们更好地理解细胞表面基因在DEGs和其他基因中的交互作用。图F.4A展示了包含1321个节点和1767条边界的PPI交联网络,该交联网络基于许多已经被验证的特征包括功能实验,共表达分析,文本挖掘,相邻区域,基因融合和数据库。我们识别出87个簇基因模式并且最相关的簇拥有超过30个交互作用的基因包括VCAM1, EGFR, TGFBR1, CD44, NGFR, ITGB2, DCC, PTPRJ, ANBCA1, HLA-DRA, CCR5 and CSF1R等。我们将之前得到的14个交叠基因与和PPI网络中至少20个交互边界的基因交叠,得到6个具有高置信度GBM表面组标志基因(图F.4B)。(这里需要强调,这里我们的数据整合了迄今为止的大量样本。)单细胞RNA测序已经证实GBM微环境内不同细胞间的共表达和相互作用驱动GBM细胞的致癌性(发生)。该研究还将GBM微环境进一步分层为巨噬细胞,少突细胞,T细胞和恶性细胞。上述得到的6个高置信基因中,只有EGFR与恶性细胞强相关,其他基因则与巨噬细胞具有强相关性。
验证高置信度GBM表面组基因和生存表达的相关分析
利用上述得到的6个差异基因,我们根据单个基因表达进行生存分析,发现6个基因的总体生存期与预后均在统计学上无意义。对数据分析,我们发现该临床数据的生存时间端点不适合总体生存期而更适合使用无进展生存期来表征。使用无进展生存期后,我们发现CD44,PTPRJ和HLA-DRA的高低表达具有统计学意义。
接下来对所有的6个基因进行生存分析,无论时OS还是DFS均无统计学意义。对CD44,PTPRJ和HLA-DRA的生存分析表明,仅在DFS上,CD44,PTPRJ和HLA-DRA的高表达与较差的预后相关。
CD44的共表达网络
OMICS研究表明,CD44在许多类型的癌症中都有过表达的现象。基于我们上述的分析,CD44在转录组和蛋白组扮演者重要角色,同时其高表达也表征更差的预后。除此之外,单细胞测序也证实CD44被富集在GBM间叶样细胞中,以及在原位异种移植的免疫缺陷小鼠中可以富集到。因此我们使用转录组数据构建CD44共表达网络进行分析。如图F.5A所示,筛选皮尔森相关系数大于0.75的边界构建CD44共表达分析网络。共有27个基因与CD44关联,具有极高相关性的基因有ELK3, CLIC4, GALNT2, TNC,和VIM。如图F.5B所示,所有的CD44共表达簇在GBM中具有高表达量(相对于正常脑组织)。结果表明CD44可能支持GBM的病理发生。
Conclusion
总之,我们使用RNA-seq数据识别了GBM表面组基因。通过一体化multi-OMICS策略,我们得到了6个GBM表面富集基因,并且这些基因在GBM发展中起重要作用。另外的,更深入的研究这些基因可能得到潜在的GBM诊断/预后标志物或GBM的治疗方案。
Methods
Data
本文分析使用了TCGA-GBM和GTEx normal brain read count 数据。最终,使用了来自TCGA的166个GBM样本和408个来自GTEx的正常脑组织数据。RNA-seq数据在预处理阶段涉及数据过滤和数据标准化。标准化所有数据集采用log2-counts per million。不可靠以及错误数据采用方差过滤(小于15)和低丰度(小于4)删除。
差异基因分析
差异分析采用NetworkAnalyst进行分析。NetworkAnalyst平台基于R包limma寻找差异基因。本文研究设置基因调整p值(adjusted p-value)小于0.05并且log2 fold change |2|。
功能富集
在DAVID 进行KEGG和GO的通路富集。
识别hub基因
利用IMEx 相互作用组学数据库对DEGs进行PPI分析。
CD44共表达分析
将原始的read counts文件存储为“.expression”文件在Graphia Prefessional平台进行共表达分析。在Graphia Prefessional处理之后,构建成对的皮尔森相关矩阵,进行每个基因表达谱基因与基因间的比较。将皮尔森相关系数大于0.7(r>0.7)存储为".pearson"文件,根据用户定义的r>0.75,构建图形网络。